Mac 499 – Trabalho de Formatura Supervisionado
2º semestre de 2001
Professor responsável: Carlos Eduardo Ferreira
Professor orientador: Marco Dimas Gubitoso
Aluna: Marina Andretta
Monografia
0.
Introdução
O projeto de Iniciação Científica a ser descrito nesta monografia é o SGEN (Simulator for Gene Expression Networks). O trabalho está dividido em quatro partes, descritas a seguir.
Na primeira parte é apresentado o projeto num nível abstrato, mostrando-se o problema de representação de uma rede de genes e a idéia de modelá-la como um sistema dinâmico discreto e estocástico.
Na segunda parte é apresentado o software construído a partir das idéias apresentadas na primeira parte, explicando-se detalhes mais concretos, como sua estrutura, a especificação da entrada e saída, etc.
Na terceira parte mostra-se como a implementação do projeto aconteceu, como foi feita a divisão do trabalho do grupo, como foram supridas novas necessidades de funcionalidade no decorrer da implementação, etc.
Na quarta e última parte são apresentadas minhas impressões pessoais sobre o desenvolvimento e implementação do SGEN, experiências obtidas e a importância do curso de graduação em Ciência da Computação para a minha participação no projeto.
1.
O
projeto
1.1.
Contextualização
Nos últimos anos, tem sido uma tendência a união da biologia, matemática e computação para a solução de problemas biológicos reais. Fenômenos biológicos, principalmente ligados à genética, têm sido modelados matemática e computacionalmente (principalmente devido à grande quantidade de dados envolvidos) com êxito e um caso interessante, que tem nessa união de disciplinas um futuro promissor é o do controle genético do ciclo da célula.
Dados e conjecturas atuais indicam que uma complexa rede de genes controla a programação de expressões genéticas que são responsáveis pelo ciclo da célula. Para acessar experimentalmente esta rede genética, o controle do ciclo da célula deve ser analisado como um sistema dinâmico integrado. A tecnologia de DNA-microarray é suficiente para servir de base para esta abordagem. Esta tecnologia permite observar padrões temporais de ativação e inibição de centenas de genes durante o ciclo da célula. Entretanto, para descobrir a rede genética (ou redes genéticas) por trás dos complexos padrões temporais da expressão dos genes, observadas através do DNA-microarray, não é uma tarefa trivial.
Sob o ponto de vista matemático, uma rede de genes pode ser modelada como um sistema dinâmico estocástico, onde cada projeção do vetor de estados representa a expressão de uma dado gene. A busca por sistemas dinâmicos que modelam redes de genes a partir de dados de DNA-microarray é um problema não-linear de identificação de sistema. Uma técnica complementar para o entendimento de sistemas dinâmicos é a simulação de um sistema definido.
No entanto, esta abordagem possui fortes limitações: o vetor de estados tem dimensão muito grande e o número de microarrays disponíveis é relativamente pequeno, o que implica em grandes erros de estimação. A maneira de superar estes problemas é acrescentar outros conhecimentos existentes sobre a rede estudada. Na prática, isso pode ser feito através do desenvolvimento de um modelo heurístico para simulações interativas e da aplicação de técnicas de identificação para melhorar o modelo. Portanto, a simulação tem um papel fundamental em pesquisas sobre redes de genes.
O SGEN (Simulator for Gene Expression Networks), desenvolvido e implementado na BIOINFO-USP (Laboratório de Bio-Informática do IME – USP), é um simulador no qual redes genéticas são representadas por sistemas dinâmicos estocásticos que possui tempo discreto e intervalos de valores de expressões também discretos (o que inclui redes Booleanas).
1.2.
Sistemas
dinâmicos
Um sistema dinâmico é representado por um vetor de funções de transição que descreve a evolução no tempo de um vetor de variáveis, chamado estado. A função de transição pode ser controlada por um sinal independente, chamado de entrada. Em geral, o que é observado não é o vetor de estados, mas sua transformação (que pode ser controlada pela entrada). Essa transformação é chamada de função de saída e o vetor resultante é chamado de saída.
Uma cadeia é um conjunto completamente ordenado. Por exemplo, um intervalo de números inteiros é uma cadeia. Um sistema dinâmico baseado em cadeias é um sistema dinâmico cujos estados são vetores de elementos de uma cadeia. Exemplos de sistemas dinâmicos baseados em cadeia são os sistemas puramente discretos, isto é, aqueles que possuem tanto tempo como intervalo de valores discretos. Uma conhecida família de sistemas puramente discretos são as redes Booleanas. Neste caso, os estados são vetores de variáveis Booleanas.
A função de transição e a função de saída são representadas por fórmulas parametrizadas. Quando estes parâmetros mudam de uma maneira estocástica, o sistema é chamado de sistema dinâmico estocástico.
1.3.
Representação
do gene
Cada gene possui um valor associado a si, que representa sua expressão no ciclo da célula. Este número pode ser obtido como uma função de valores associados a outros genes (ou ao próprio gene) em instantes anteriores, chamada função de transição.
Como estamos lidando com um sistema estocástico, a função que define o valor de um gene não é única: a cada instante de tempo, uma função representa um gene com uma certa probabilidade.
Por exemplo, digamos que temos os genes G1, G2, G3 e G4. Suponha que temos os valores dos graus de ativação destes quatro genes nas cinco unidades de tempo anteriores:
|
Unidades de tempo transcorridas (delay) |
G1 |
G2 |
G3 |
G4 |
|
1 |
0 |
3 |
-2 |
1 |
|
2 |
0 |
2 |
0 |
1 |
|
3 |
0 |
4 |
1 |
1 |
|
4 |
1 |
0 |
1 |
1 |
|
5 |
2 |
-1 |
0 |
1 |
O valor do grau de ativação de G1 no instante atual (denotado por G1[0]) é uma função j de G2 há 3 unidades de tempo (denotado por G2[3]) e de G4 há 1 unidade de tempo (denotado por G4[1]), com probabilidade de 0.25. Ou seja, com 25% de chances, G1[0] = j (G2[3], G4[1]). Com probabilidade 0.75, G1[0] = j (G2[3], G4[2]).
Note que são necessários alguns dos valores assumidos anteriormente por cada gene. Mais precisamente, para cada Gi, são necessários todos os valores de Gi[1] a Gi[k], onde k é o maior delay associado a Gi dentre todas as funções definidas, ou seja, k = Max{j: Gi[j] aparece como argumento de alguma função de transição do sistema}.
1.4.
Representação
da função de transição
A função que associa um valor a um gene pode ser vista como uma matriz de dimensão n, onde n é o número de argumentos desta função. Cada dimensão da matriz está associada a um argumento e, portanto, representa os valores do domínio deste argumento. No interior da matriz estão os valores que a função de transição assume para cada n-upla de valores.
Para ilustrar, vejamos a representação da função de transição j do exemplo anterior:
|
|
-5 |
-4 |
-3 |
-2 |
-1 |
0 |
1 |
2 |
3 |
4 |
5 |
|
-5 |
|
|
|
|
|
|
|
|
|
|
|
|
-4 |
|
|
|
|
|
|
|
|
|
|
|
|
-3 |
|
|
|
|
|
|
|
|
|
|
|
|
-2 |
|
|
|
|
|
|
|
|
|
|
|
|
-1 |
|
|
|
|
|
|
|
|
|
|
|
|
0 |
|
|
|
|
|
|
|
|
|
|
|
|
1 |
|
|
|
|
|
|
|
|
|
|
|
|
2 |
|
|
|
|
|
|
|
|
|
|
|
|
3 |
|
|
|
|
|
|
|
|
|
|
|
|
4 |
|
|
|
|
|
|
|
|
|
|
|
|
5 |
|
|
|
|
|
|
|
|
|
|
|
As linhas representam os valores do primeiro argumento e as colunas representam os valores do segundo argumento. As posições em cinza escuro assumem valor 1, as posições em cinza claro assumem 0 e as posições em branco assumem -1. Assim, j (-4, 0) = 1, por exemplo.
No geral há uma grande proximidade das posições que assumem valores iguais, então é interessante a representação da função de transição não como uma matriz, mas como uma árvore de intervalos, em que cada nível representa um argumento e cada nó corresponde a um intervalo do domínio.
No exemplo da função de transição j descrita pela matriz acima, uma árvore possível seria:
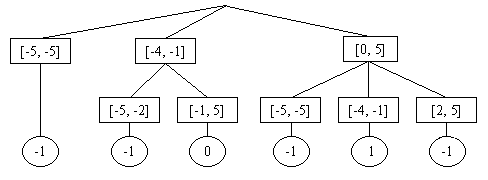
Note que, dada uma n-upla, para calcular o valor da função de transição basta percorrer a árvore da seguinte maneira:
a) procurar, no primeiro nível da árvore, o nó com o intervalo que contém o primeiro argumento;
b) a partir deste nó, procurar nos seus nós filhos o intervalo que contém o segundo argumento;
c) repetir o passo (b) para todos os argumentos restantes;
d) o valor da função de transição é dado pelo último nó alcançado (representado por circunferências no diagrama acima).
Nos casos em que não há grande proximidade das posições que assumem valores iguais, é muito trabalhoso descrever uma árvore como esta. Nestes casos, a descrição da função de transição como uma expressão é mais interessante.
1.5.
A
simulação
Para simular o comportamento de uma rede de genes no tempo são necessárias as definições dos genes da rede, isto é, seus valores em unidades de tempo passadas (denominado histórico do gene), as dependências existentes entre os genes da rede (ou seja, qual a função de transição, quais os argumentos da mesma e com que probabilidade cada função é escolhida) e as definições das funções de transição (como descrito em 1.4).
A partir desses dados, a simulação da evolução dos genes em uma unidade de tempo consiste em:
a) para cada gene, sortear a função de transição f para ser usada, dada sua probabilidade de ser escolhida;
b) calcular o valor de f dados os valores dos argumentos no instante atual;
c) atualizar o histórico de cada gene com os valores recém calculados. Isto significa que todos os valores de delay i passam a representar o valor do gene de delay i + 1 (pois mais uma unidade de tempo se passou) e o novo valor calculado passa a ser o valor de delay 1.
Para ficar mais claro, sejam os genes definidos em 1.3 e a função de transição j definida em 1.4. Já temos definido que G1[0] = j (G2[3], G4[1]) com probabilidade 0.25 e G1[0] = j (G2[3], G4[2]) com probabilidade 0.75. Ainda precisamos definir G2[0], G3[0] e G4[0]. Suponha que G2[0] = j (G2[3], G3[2]), G3[0] = j (G1[1], G2[1]) e G4[0] = j (G4[2], G4[5]), todos com probabilidade 1. Simulando a evolução deste sistema em 1 unidade de tempo temos que:
G1[0] = j (G2[3], G4[1]) = j (4, 1) = -1;
G2[0] = j (G2[3], G3[2]) = j (4, 0) = -1;
G3[0] = j (G1[1], G2[1]) = j (0, 3) = 1;
G4[0] = j (G4[2], G4[5]) = j (1, 1) = 1;
Como avançamos o sistema em 1 unidade de tempo, atualizamos os históricos dos genes:
|
Unidades de tempo transcorridas (delay) |
G1 |
G2 |
G3 |
G4 |
|
1 |
-1 |
-1 |
1 |
1 |
|
2 |
0 |
3 |
-2 |
1 |
|
3 |
0 |
2 |
0 |
1 |
|
4 |
0 |
4 |
1 |
1 |
|
5 |
1 |
0 |
1 |
1 |
Note que, para observar a evolução do sistema em t unidades de tempo, basta repetir este processo t vezes.
2.
O
software
O SGEN (Simulator for Gene Expression Networks) é um programa feito basicamente em linguagem C, que simula uma rede de genes modelada como um sistema dinâmico discreto e estocástico.
Atualmente existem duas versões: uma usa as funções de transição genéricas, que podem assumir qualquer valor inteiro e têm a representação em forma de árvore como a descrita na seção 1.4.
A outra usa funções de transição booleanas, nas quais a matriz que representa a função é um reticulado booleano. Assim, verificar se um argumento a pertence a um intervalo [i, f] significa verificar se o sup(a, i) = a e sup(a, f) = f.
2.1.
A
estrutura
O fluxo de execução do SGEN segue o diagrama a seguir:
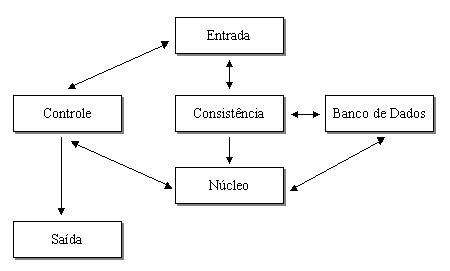
2.2.
O
controle
O controle é a interface entre o usuário e o próprio Simulador. Através de uma espécie de shell, o usuário tem a possibilidade de carregar os dados para a simulação, simular a evolução do sistema em tantos passos quanto queira, editar os históricos dos genes existentes, acrescentar/remover novos genes ou funções ao/do sistema, salvar os dados e visualizar os resultados obtidos.
2.3.
A
entrada
A entrada do SGEN é feita na forma de texto. O Controle permite que os dados sejam inseridos via arquivo ou via prompt (do Controle).
A seguir são apresentadas as sintaxes de todos os dados de entrada exigidos pelo Simulador e alguns exemplos. Caracteres em negrito devem aparecer na entrada exatamente da mesma maneira com são apresentados aqui. Itens em itálico devem ser substituídos de acordo com a explicação que segue cada descrição de entrada. Comentários devem ser iniciados com #.
2.3.1.
Funções
de transição
2.3.1.1.
Como
árvores
A função de transição é descrita pela seguinte sintaxe:
def nome: lista de Intervalos;
Onde:
nome é uma string que representa o nome da função de transição; e
lista de Intervalos é uma seqüência de Intervalos, separados por espaços em branco, onde Intervalos é dado por:
[início, fim]: valor ou
[início, fim]: Intervalo
Onde:
início é um número inteiro que representa o início do intervalo;
fim é um número inteiro que representa o fim do intervalo; e
valor é um número inteiro que representa o valor que a função assume neste intervalo.
Para o exemplo da seção 1, teríamos os genes definidos da seguinte maneira:
def Fi:
[-5, -5]: -1
[-4, -1]:
[-5, -2]: -1
[-1, 5]: 0
[0, 5]:
[-5, -5]: -1
[-4, 1]: 1
[2, 5]: -1
;
2.3.1.2.
Como
expressões
A sintaxe das expressões é a seguinte:
expr nome: expressão;
Onde:
nome é uma string que representa o nome da expressão; e
expressão é uma string que representa a expressão de cálculo da função de transição. Esta expressão pode conter os seguintes comandos:
op1 op2 + : soma op1 e op2;
op1 op2 - : subtrai op2 de op1;
op1 op2 * : multiplica op1 e op2;
op1 op2 / : divide op1 por op2;
op1 op2 > : verdadeiro se op1 maior que op2;
op1 op2 < : verdadeiro se op1 menor que op2;
op1 op2 = : verdadeiro se op1 igual a op2;
op1 op2 != : verdadeiro se op1 diferente de op2;
op1 op2 >= : verdadeiro se op1 maior ou igual a op2;
op1 op2 <= : verdadeiro se op1 menor ou igual a op2;
op1 op2 = : verdadeiro se op1 igual a op2;
$n : retorna argumento n;
!n : armazena argumento n;
@n : recupera argumento n;
SUM n1 n2 : retorna a soma dos argumento n1 até n2;
ret : retorna o valor da expressão;
if (expressão booleana) expr1 else expr2; : calcula expr1 se expressão booleana for verdadeira e expr2 caso contrário;
in início fim n : retorna verdadeiro se n está entre início e fim;
while (expressão booleana) expressão; : calcula expressão enquanto expressão booleana for verdadeira;
Exemplo:
expr decr: $1 30 - !1 if (@1 -100 <) -100 !1; @1 ret;
2.3.2.
Genes
O gene é descrito pela seguinte sintaxe:
Gene nome: {
Probabilidade NomeDaFunção ListaDeArgumentos;
...
};
Onde:
nome é uma string que representa o nome do gene;
Probabilidade é um número real entre 0 e 1 que representa a
probabilidade com a qual a função NomeDaFunção deve ser calculada o novo
valor do gene;
NomeDaFunção é uma string que representa o nome da função de transição; e
ListaDeArgumentos são os argumentos da função NomeDaFunção. Os argumentos seguem a seguinte sintaxe:
gene[delay] ... gene[delay]
Onde:
gene é um nome de gene; e
delay é um número inteiro que representa o delay.
Para o exemplo da seção 1, teríamos os genes definidos da seguinte maneira:
Gene G1: {
0.25 Fi G2[3]
G4[1];
0.75 Fi G2[3] G4[2];
};
Gene G2: {
1.0 Fi
G2[3] G3[2];
};
Gene G3: {
1.0 Fi G1[1] G2[1];
};
Gene G4: {
1.0 Fi G4[2] G4[5];
};
2.3.3.
Históricos
O histórico de cada gene é dado pela seguinte sintaxe:
hist gene: [lista dos valores];
Onde:
gene é o nome do gene que possui o histórico descrito, e
lista de valores é uma lista de n números inteiros, separados por espaços em branco, que representa os valores de gene com delay 1, ..., n, nesta ordem.
Para o exemplo da seção 1, teríamos os históricos definidos da seguinte maneira:
hist G1: [0 0 0 1 2];
hist G2: [3 2 4 0 -1];
hist G3: [-2 0 1 1 0];
hist G4: [1 1 1 1 1];
2.4.
O
teste de consistência
Para ser possível a simulação da rede de genes, é necessário que o sistema esteja consistente, ou seja, algumas restrições devem ser satisfeitas:
- para um dado gene, as probabilidades de cada função de transição a ser escolhida deve entre 0 e 1 e a soma das probabilidades de todas estas funções deve ser igual a 1;
- os históricos dos genes devem ser suficientemente completos para o cálculo da evolução do sistema, ou seja, para um gene g, o histórico de g deve conter k valores, onde k é o maior delay que aparece associado a g em toda a definição do sistema;
- quando um nome de gene aparece como argumento de alguma função, um gene com este nome deve ser definido;
- quando um nome de função aparece na definição de um gene, uma função com este nome deve ser definida.
2.5.
O
banco de dados
O banco de dados é uma tabela de hash que armazena todas as estruturas necessárias para a simulação (as funções e os genes com seus históricos). A partir desta tabela é possível verificar a consistência de todo o sistema cada vez que uma nova função ou um novo gene é inserido, removido ou editado. Pode-se também optar por ignorar alguns genes em uma simulação ou até mesmo alterar a definição da função de transição de um gene sem precisar redefinir a função (quando ela já pertence ao banco de dados).
Este banco de dados também permite salvar todo o sistema ou parte dele (na forma de texto, como a entrada).
2.6.
O
núcleo
O núcleo é responsável pela simulação em si. Na realidade esta é a parte mais simples do simulador.
Como já foi verificado que os dados são consistentes, basta percorrer todos os genes, sortear a função de transição para cada um deles, calcular o valor da função e atualizar o histórico de cada um deles. Este procedimento é repetido tantas vezes quantas o controle solicitar.
2.7.
A
saída
Atualmente existem dois tipos de saída para a simulação: uma em formato texto e outra em formato gráfico.
A saída em formato texto nada mais é do que a impressão de cada nova linha do histórico dos genes calculada a cada passo da simulação.
No formato gráfico, apresenta-se a evolução dos genes através de um gráfico de linhas, colocando-se o tempo transcorrido no eixo das abscissas e os valores dos genes no eixo das ordenadas. Cada linha representa um gene.
3.
O
grupo de trabalho e a implementação
O grupo responsável pelo SGEN foi criado em abril de 2000. No início ele era composto pelos alunos do Bacharelado em Ciência da Computação Marina Andretta, Fábio Massaaki Katayama, Fabiana Guarsoni Rocha, Rafael Pereira Luna e Ricardo Hideo Sahara, orientados pelos professores Dr. Marco Dimas Gubitoso e Dr. Júnior Barrera.
Nesta primeira fase foram feitas reuniões semanais para que os dois professores explicassem as motivações do projeto, a modelagem da rede de genes e as idéias para implementação do simulador. Alguns pontos da modelagem ainda estavam em aberto, então todo o grupo participou de discussões do modelo.
Com o modelo definido, os alunos foram divididos em dois grupos: Marina, Fábio e Fabiana responsáveis pela implementação do núcleo e das estruturas internas das funções e genes em linguagem C; e Rafael e Ricardo responsáveis pelo tratamento da entrada e saída (usando analex) e implementação do banco de dados (tabela de símbolos implementada como tabela de hash) em linguagem C. Tudo sob a orientação do professor Dr. Marco Dimas Gubitoso.
Após o início da implementação, Fabiana deixou o projeto e Edgar Szilagy passou a fazer parte do sub-grupo do Rafael e do Ricardo. Algum tempo depois, Ricardo também deixou o projeto.
Com o passar do tempo, com uma primeira versão do simulador funcionando, foram-se tornando necessárias a inclusão de algumas funcionalidades, como inclusão do elemento estocástico nas funções, a possibilidade de edição dos genes e funções, a descrição de funções como expressões, saída em forma de gráfico, entre outras. Cada vez que surgia uma nova necessidade, ela era incluída por algum componente do grupo (os alunos ou o próprio professor) no Simulador. Isso fez com que o código, antes razoavelmente modularizado, passa-se a ficar cada vez mais confuso e remendado.
Por isso, a idéia agora é a de reformular o código, tornando-o orientado a objeto (implementado em C++) e possivelmente paralelizado. Pretende-se também incluir a possibilidade de apresentação dos dados de simulação de várias maneiras gráficas diferentes, bem como diversas maneiras de representação da entrada.
4.
Experiência
pessoal
Este projeto de Iniciação Científica foi uma experiência fantástica para mim. Trabalhar num projeto desde sua elaboração até o uso do software produzido é muito interessante e serve como base prática para a elaboração de novos projetos.
Tive a oportunidade de participar de todos os passos para a elaboração e implementação do projeto. Pude observar como surgem as idéias para a modelagem do problema proposto, a discussão dessa modelagem (O fenômeno biológico está bem representado? A modelagem escolhida é correta e genérica o suficiente para as necessidades apresentadas?), como pontos do modelo mudam no decorrer do tempo, a divisão de tarefas no grupo de trabalho, a escolha da linguagem de programação, as tentativas de implementação na linguagem escolhida, as adaptações que precisam ser feitas a pedido do usuário final e até mesmo a generalização do software para uso em outras áreas que podem ser modeladas de maneira similar.
Além do aprendizado prático citado acima, tive a oportunidade de aprender um pouco de teoria, como sistemas dinâmicos discretos e estocásticos e sua aplicabilidade em bioquímica e em outras áreas.
Alguns cursos da graduação em Ciência da Computação foram fundamentais para a concepção e implementação do SGEN. Dentre eles estão:
- Estruturas de Dados (mac323): talvez o curso de maior importância, pois tiveram de ser construídas árvores para a representação das funções, listas ligadas das estruturas que representam os genes, tabela de hash para armazenar todos os dados para a simulação (genes e funções) e um vetor de vetores circulares para a simulação (além de outras estruturas secundárias);
- Conceitos Fundamentais de Linguagens de Programação (mac316): a construção de uma gramática para a representação dos dados de entrada (funções, genes e históricos de cada gene), a transformação dos dados lidos em forma de texto (seguindo a gramática especificada) numa representação interna e o armazenamento desta representação numa tabela de símbolos são características típicas da implementação de uma linguagem de programação;
- Álgebra Booleana e Aplicações (mac329): um caso especial e muito importante de função para descrição do estado dos genes envolve reticulados booleanos;
É claro que os cursos de Introdução à Computação (mac110), Princípios de Desenvolvimento de Algoritmos (mac122), Laboratório de Programação I (mac211) e Laboratório de Programação II (mac242) também foram de vital importância para que eu tivesse uma boa base para a implementação do SGEN. Estes cursos não ajudaram em nenhum ponto específico (por serem cursos introdutórios), mas me deram a base de programação e construção de algoritmos necessária para que eu fosse capaz não só de criar e implementar partes do SGEN, mas de compreender conceitos necessários para assimilar os cursos mais avançados citados acima.